Computational Design of an α-Gliadin Peptidase
Página 1 de 1.
 Computational Design of an α-Gliadin Peptidase
Computational Design of an α-Gliadin Peptidase
ABSTRACT
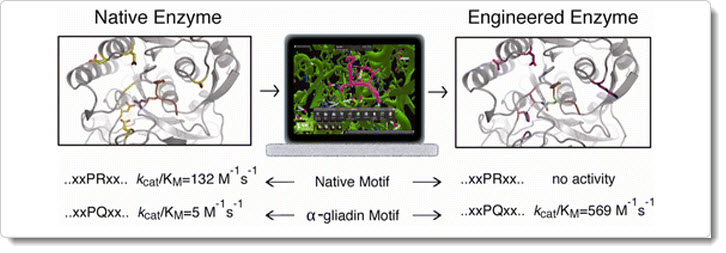
The ability to rationally modify enzymes to perform novel chemical transformations is essential for the rapid production of next-generation protein therapeutics. Here we describe the use of chemical principles to identify a naturally occurring acid-active peptidase, and the subsequent use of computational protein design tools to reengineer its specificity toward immunogenic elements found in gluten that are the proposed cause of celiac disease. The engineered enzyme exhibits a kcat/KM of 568 M−1 s−1, representing a 116- fold greater proteolytic activity for a model gluten tetrapeptide than the native template enzyme, as well as an over 800-fold switch in substrate specificity toward immunogenic portions of gluten peptides. The computationally engineered enzyme is resistant to proteolysis by digestive proteases and degrades over 95% of an immunogenic peptide implicated in celiac disease in under an hour. Thus, through identification of a natural enzyme with the pre-existing qualities relevant to an ultimate goal and redefinition of its substrate specificity using computational modeling, we were able to generate an enzyme with potential as a therapeutic for celiac disease.
Sydney R. Gordon, Elizabeth J. Stanley, Sarah Wolf, Angus Toland, Sean J. Wu, Daniel Hadidi, Jeremy H. Mills, David Baker, Ingrid Swanson Pultz, and Justin B. Siegel
Journal of the American Chemical Society 2012 134 (50), 20513-20520
Journal of the American Chemical Society 2012 134 (50), 20513-20520
Fuente: ACS Publications
 Temas similares
Temas similares» Industrial Design by Günter Kupetz
» Introduction to Autocad 2010 2D and 3D Design by Alf Yarwood
» RCSTR + Design Spec ♦ Tutorial Aspen Plus
» Analysis, Synthesis, and Design of Chemical Processes
» Design of Experiments Practical Guide by Paul D. Funkenbusch
» Introduction to Autocad 2010 2D and 3D Design by Alf Yarwood
» RCSTR + Design Spec ♦ Tutorial Aspen Plus
» Analysis, Synthesis, and Design of Chemical Processes
» Design of Experiments Practical Guide by Paul D. Funkenbusch
Página 1 de 1.
Permisos de este foro:
No puedes responder a temas en este foro.
 Índice
Índice
